Podstawowe pojęcia genetyczne, DNA
DNA, czyli kwas deoksyrybonukleinowy to materialny nośnik kodu genetycznego odpowiedzialnego za budowę i sposób funkcjonowania organizmu, czyli o ogromnej ilości jego cech. Każda taka cecha, o ile jest oczywiście cechą dziedziczną, zapisana jest w genach – fragmentach DNA. Nie obowiązuje tu zasada jeden gen – jedna cecha. Są cechy, które są efektem obecności kilku genów, jak również zdarza się, że jeden gen wywołuje pojawienie się kilku cech jednocześnie. Liczba genów człowieka to ponad 20 tysięcy, a średnia długość genu to około 27 tysięcy par zasad. Łączna wielkość genomu człowieka to 3,2 miliarda par zasad a jego długość wynosi ok. 100 cm. Odpowiada to 800 MB informacji.
 Większość informacji genetycznych jest zawarta w chromosomach, a pozostałe znajdują się w mitochondriach.
Większość informacji genetycznych jest zawarta w chromosomach, a pozostałe znajdują się w mitochondriach.
Chromosomy są niewielkimi strukturami w jądrze komórkowym, w których zawarta jest większość materiału genetycznego człowieka DNA. Mamy 23 pary chromosomów: autosomalne (chromosomy 1-22) i heterosomalne (X i Y) i te ostatnie decydują o płci. Mężczyźni posiadają chromosomy XY, a kobiety XX. Dziedziczenie chromosomu Y jest bardzo proste – przechodzi on wyłącznie z ojca na syna (YDNA). Mężczyźni dziedziczą także chromosom X po matce. Kobiety dziedziczą chromosom X po ojcu i jeden X po swojej matce.
Pozostała informacja genetyczna jest w mitochondriach znajdujących się poza jądrem komórkowym. DNA mitochondrialny (mtDNA) jest dziedziczony wyłącznie po matce. Geny mitochondrialne są przekazywane przez matkę potomstwu obu płci.
W wyniku rozwoju genetyki powstały nowe dziedziny badawcze: genealogia genetyczna, genetyka populacyjna, archeologia genetyczna, genetyka sądowa itp. Badając problem pochodzenia ludzi współczesnych posługujemy się m.in. dwoma bardzo wygodnymi narzędziami – zmiennością mitochondrialnego DNA (mtDNA) i chromosomu Y. Powstające w nich niewielkie zmiany (mutacje) są przekazywane z pokolenia na pokolenie. Na ich podstawie można ustalić pokrewieństwo ludzi, czyli określić wspólnych przodków i ustalić orientacyjny czas, kiedy oni żyli.
Mitochondrialna Ewa – w genetyce hipoteza o istnieniu kobiety, od której pochodzą współcześni ludzie. W wyniku porównywania DNA mitochondriów pochodzących od różnych ludzi stwierdzono, że każdy żeński rodowód można ostatecznie sprowadzić do pojedynczej kobiety, która żyła w Afryce 140–240 tys. lat temu. Kobiety tej nie należy uważać za pramatkę ludzkości, bo współcześnie z nią żyło jeszcze wiele innych kobiet i mężczyzn, po których dziedziczymy geny jądrowe.
Y-chromosomalny Adam – hipoteza zakładająca, że istniał ostatni wspólny przodek w linii ojcowskiej, od którego pochodzą współcześni ludzie. Współcześni Y-chromosomalnemu Adamowi oraz część żyjących przed nim, mieli inny Y-chromosom, ale ich rody z biegiem czasu w linii męskiej wymarły lub jeszcze nie zostały odkryte.
Haplogrupa (Hg) to grupa podobnych genów w organizmie, które są dziedziczone razem od jednego rodzica i które mają wspólnego przodka z ta samą mutacją. Haplogrupy dzielą się na dodatkowe podgrupy (subclades). W genetyce populacji posługujemy się haplogrupami męskimi (haplogrupa Y, w skrócie Hg YDNA) oraz żeńskimi (haplogrupa mitochondrialna, w skrócie Hg mtDNA).
Testy genetyczne
- autosomalny DNA – zawiera najwięcej informacji o naszych przodkach, gdyż dziedziczymy go po wszystkich przodkach i wielu wcześniejszych pokoleniach; umożliwia określenie pokrewieństwa u osób z różnymi haplogrupami YDNA oraz mtDNA
- YDNA – pozwala na prześledzenie wyłącznie bezpośredniej męskiej linii przodków i ustalenie męskiej haplogrupy (test może być wykonany tylko przez mężczyzn)
- DNA mitochondrialny – zawiera informacje o naszej bezpośredniej żeńskiej linii przodków i zbadanie go pozwala na ustalenie mitochondrialnej haplogrupy (test może być wykonany przez każdą osobę bez względu na płeć).
Test autosomalny atDNA
Głównym celem testu autosomalnego DNA jest dopasowanie do innych osób. Tam, gdzie badana osoba ma kilka kolejnych wspólnych SNP z inną testowaną osobą w bazie danych firmy wykonującej testy, można wywnioskować, że dzielą one segment DNA w tej części swoich genomów. Jeśli segment jest dłuższy niż kwota progowa ustalona przez firmę testującą, te dwie osoby uważa się za pasujące. Algorytmy używane do ustalenia dopasowania, są zastrzeżone i specyficzne dla każdej firmy.
Uwaga! Firmy na tej podstawie podają pokrewieństwo – kuzyni 3, 4, 5,… rzędu, co oczywiście jest tylko prawdopodobieństwem a nie pewnikiem.
Jednostką dla segmentów DNA jest centimorgan (cM). Dla porównania, pełny ludzki genom wynosi około 6500 cM. Im krótsza długość porównania, tym większe prawdopodobieństwo, że porównanie jest fałszywe. Ważną statystyką dla późniejszej interpretacji jest długość wspólnego DNA (lub procent dzielonego genomu).
Interpretacja dopasowań atDNA
Większość firm pokazuje klientom, ile cM współdzieli i ile segmentów. Z liczby cM i segmentów można oszacować związek między dwoma osobami, jednak ze względu na losowy charakter dziedziczenia DNA, szacunki związku, szczególnie dla odległych krewnych, są jedynie przybliżone. Niektórzy dalecy kuzyni w ogóle się nie dopasują. Kluczową informacją jest procent DNA dzielony przez 2 osoby. Może to wskazywać na bliskość związku. Jednak nie pokazuje on ról dwóch osób – np. 50% wspólnych genów sugeruje związek rodzic – dziecko, ale nie określa, która osoba jest rodzicem.
Haplogrupa YDNA – oznaczenie
Haplogrupa autora tekstu (vayda) to I2a1b2a1a1a1a2, gdzie litera I oznacza główną haplogrupe, a kolejne cyfry i litery to mutacje (sublades) haplogrupy I. Te mutacje wystąpiły w okresie od 43 tysiące lat temu do roku 550 naszej ery. Oznaczenie jest według systemu ISOGG, ale stosuje się również nazwy markerów SNP i w tym wypadku jest to A2423 lub Y12911 zależnie od laboratorium wykonującego testy DNA.
Genealogia genetyczna
Wiele osób poszukuje swoich krewnych wykorzystując wyniki DNA i portale takie jak FamilyTreeDNA (FTDNA), które szukają dopasowań. Trzeba jednak brać pod uwagę, że obecnie baza danych tych firm jest niewielka i znalezienie krewnych ma małe szanse. W Polsce jest przebadanych jedynie kilka tysięcy osób. Dla niektórych krajów jak Szkocja, Irlandia, Norwegia czy Anglia jest już dużo wyników i tam szanse są większe. W praktyce ludzie znajdują krewnych, a niektóre „młode” mutacje są identyfikowane z konkretnymi nazwami rodów/klanów.
Formed time i TMRCA
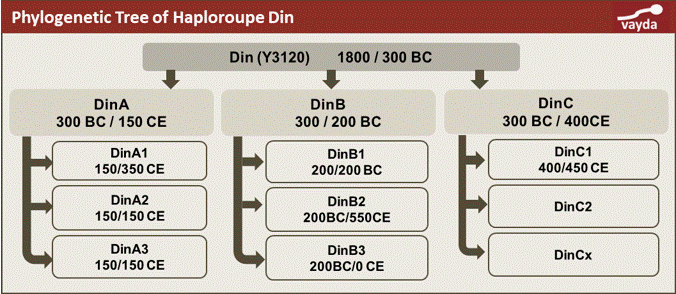
W określaniu wieku haplogrupy używa się dwóch okresów Formed time (czas postania mutacji) i TMRCA (Time to Most Recent Common Ancestor – czas do najbliższego wspólnego przodka). Poniżej rysunek wyjaśniający – pokazuje linie pokoleń dla mutacji haplogrupy nazwanej Din oraz kolejnych późniejszych mutacji. Czas i kolejność ich pojawienia się są hipotetyczne, bowiem nie wynika to z ich przybliżonych dat formed i TMRCA, ale o tym za chwile.

Mutacja dla Hg Din wystąpiła 1800 lat pne, a TMRCA wynosi 300 pne. To oznacza, że najbliższy wspólny przodek – mający mutacje Din – dla populacji Din oraz populacji z kolejnymi mutacjami DinA, DinB itd. żył 300 lat pne. Jest to czas, gdy wystąpiła najwcześniejsza mutacja, czyli DinA.
Czas Formed i TMRCA to nie jest precyzyjna liczba lat temu, a średnia z zakresu. Na przykład:
- haplogrupa A0T: formed 235900 (243700-228300), TMRCA 1613000 (169900-152900), dokładność +/- 8000 lat
- haplogrupa I: formed 42900 (45700-40300), TMRCA 27500 (29800-25200), dokładność +/- 2500 lat
- haplogrupa A2423: formed 1450 (1750-1100), TMRCA 750 (1350-400), dokładność +/- 500 lat
To uśrednianie często powoduje fałszywe lub niepewne wnioski przez osoby, które nie zagłębiły się w temat. Wyjaśnię to na poniższym przykładzie drzewa mutacji (ang. Phylogenetic tree).
To drzewo przedstawia haplogrupe Din oraz podgrupy (mutacje) DinA, DinB, DinC oraz ich kolejne mutacje. Przykładowo dla DinB mamy formed/TMRCA zapisany w formie 300/200 pne. Pierwsza rzecz, która zaskakuje, to że mutacje w gałęziach drzewa występują w tym samy czasie np. DinB1, DinB2, DinB3 – 200 lat pne. To jest reguła dla całego drzewa mutacji dla podgrup, ale to wynika z tego, że daty są przybliżane. W tym wypadku 200 lat pne jest średnią z zakresu lat 600 pne – 50 ne. W rzeczywistości te mutacje wystąpiły w różnym czasie w tym przedziale czasowym, tylko genetycy nie mają metody dokładnego wyznaczania.
Drugie zaskoczenie, to że TMRCA wyższej mutacji jest ten sam co czas powstania niższych mutacji np. TMRCA DinB jest tożsamy z datami powstania niższych mutacji DinB1, DinB2, DinB3 i wynosi 200 lat pne. To jest reguła dla całego drzewa mutacji. Oczywiście to także jest przybliżenie. W rzeczywistości to znów mogło się zdarzyć w przedziale lat 600 pne – 50 ne.
I wreszcie trzeci przypadek na przykładzie DinB1 gdzie czas mutacji jest ten sam co TMRCA – 200 lat pne. Owszem, może się tak zdarzyć, że mutacje występują szybko w kolejnych pokoleniach, ale to występuje rzadko. Wyjaśnia to znów zakres dat wynoszący aż 900 lat pne – 450 lat ne.
Zatem wyciąganie wniosków z takich dat na temat rozwoju populacji, bo w krótkim czasie było dużo mutacji, oraz z drugiej strony, że było ich mało to był regres populacji (ang. bottleneck – wąskie gardło), jest nieuprawnione. Do prawidłowej oceny rozwoju populacji używa się przede wszystkim genów autosomalnych, a nie Y DNA.
vayda
Dodatki
Genetycy okresy określają używając nazw epok, okresów i kultur, dlatego zamieszczam „ściąge”
 3702total visits,1visits today
3702total visits,1visits today